科еӯҰ家жҸҗеҮәж–°жЁЎеһӢ жңүеҠ©дәҺзҗҶи§ЈеҲҶеӯҗиҮӘз»„з»ҮжҲҗз”ҹе‘Ҫз»“жһ„зҡ„иҝҮзЁӢ
 (иө„ж–ҷеӣҫзүҮ)
(иө„ж–ҷеӣҫзүҮ)
дёҖдёӘж–°жЁЎеһӢжңүеҠ©дәҺзҗҶи§ЈеҲҶеӯҗиҮӘз»„з»ҮжҲҗз”ҹе‘Ҫз»“жһ„зҡ„иҝҮзЁӢгҖӮз”ҹе‘ҪеҮәзҺ°зҡ„дёҖдёӘжҪңеңЁеҒҮи®ҫж¶үеҸҠзӣёдә’е…іиҒ”зҡ„еҲҶеӯҗиҮӘз»„иЈ…жҲҗзұ»дјјз»Ҷиғһж¶Іж»ҙзҡ„з»“жһ„гҖӮиҝҷдәӣзү№е®ҡзҡ„еҲҶеӯҗз»„еҸҜд»Ҙе»әз«ӢжңҖж—©зҡ„иҮӘжҲ‘еӨҚеҲ¶д»Ји°ўеҫӘзҺҜпјҢиҝҷжҳҜз”ҹзү©зі»з»ҹжҷ®йҒҚеӯҳеңЁзҡ„дёҖдёӘзү№еҫҒпјҢеңЁжүҖжңүз”ҹе‘ҪеҪўејҸдёӯйғҪжҳҜдёҖиҮҙзҡ„гҖӮж №жҚ®иҝҷдёҖиҢғејҸпјҢжңҖж—©зҡ„з”ҹзү©еҲҶеӯҗйңҖиҰҒйҖҡиҝҮзј“ж…ўе’Ңж•ҙдҪ“ж•ҲзҺҮдҪҺдёӢзҡ„иҝҮзЁӢиҒҡйӣҶеңЁдёҖиө·гҖӮ
иҝҷз§Қзј“ж…ўзҡ„йӣҶзҫӨеҪўжҲҗдјјд№ҺдёҺз”ҹе‘ҪеҮәзҺ°зҡ„йҖҹеәҰдёҚз¬ҰгҖӮMPI-DS з”ҹе‘Ҫзү©иҙЁзү©зҗҶзі»зҡ„科еӯҰ家们зҺ°еңЁжҸҗеҮәдәҶдёҖдёӘжӣҝд»ЈжЁЎеһӢпјҢеҸҜд»Ҙи§ЈйҮҠиҝҷз§ҚйӣҶзҫӨзҡ„еҪўжҲҗпјҢд»ҺиҖҢи§ЈйҮҠеҪўжҲҗз”ҹе‘ҪжүҖйңҖзҡ„еҢ–еӯҰеҸҚеә”зҡ„еҝ«йҖҹеҸ‘з”ҹгҖӮ
иҝҷйЎ№з ”з©¶зҡ„第дёҖдҪңиҖ…ж–ҮжЈ®зү№-з“Ұиөһ-йӣ·еёғе°”пјҲVincent Ouazan-ReboulпјүиҜҙпјҡ"дёәжӯӨпјҢжҲ‘们иҖғиҷ‘дәҶдёҚеҗҢзҡ„еҲҶеӯҗпјҢеңЁдёҖдёӘз®ҖеҚ•зҡ„ж–°йҷҲд»Ји°ўеҫӘзҺҜдёӯпјҢжҜҸдёӘзү©з§ҚйғҪдјҡдә§з”ҹдёӢдёҖдёӘзү©з§ҚжүҖйңҖзҡ„еҢ–еӯҰзү©иҙЁгҖӮ"
"жЁЎеһӢдёӯзҡ„е”ҜдёҖиҰҒзҙ жҳҜеҲҶеӯҗзҡ„еӮ¬еҢ–жҙ»жҖ§гҖҒеҲҶеӯҗи·ҹйҡҸе…¶дә§з”ҹе’Ңж¶ҲиҖ—зҡ„еҢ–еӯҰзү©иҙЁжө“еәҰжўҜеәҰзҡ„иғҪеҠӣпјҢд»ҘеҸҠеҫӘзҺҜдёӯеҲҶеӯҗйЎәеәҸзҡ„дҝЎжҒҜпјҢ"他继з»ӯиҜҙгҖӮ
дёҖдёӘж–°жЁЎеһӢжҸҸиҝ°дәҶеҸӮдёҺд»Ји°ўеҫӘзҺҜзҡ„еӮ¬еҢ–еүӮзҡ„иҮӘз»„з»ҮиҝҮзЁӢгҖӮдёҚеҗҢз§Қзұ»зҡ„еӮ¬еҢ–еүӮпјҲд»ҘдёҚеҗҢйўңиүІиЎЁзӨәпјүеҪўжҲҗз°ҮзҫӨпјҢ并иғҪзӣёдә’иҝҪйҖҗгҖӮиө„ж–ҷжқҘжәҗпјҡMPI-DS / LMP
еӣ жӯӨпјҢиҜҘжЁЎеһӢжҳҫзӨәдәҶеҢ…жӢ¬еҗ„з§ҚеҲҶеӯҗзү©з§ҚеңЁеҶ…зҡ„еӮ¬еҢ–з°Үзҡ„еҪўжҲҗгҖӮжӯӨеӨ–пјҢйӣҶзҫӨзҡ„еўһй•ҝйҖҹеәҰе‘ҲжҢҮж•°зә§еўһй•ҝгҖӮеӣ жӯӨпјҢеҲҶеӯҗеҸҜд»Ҙеҝ«йҖҹгҖҒеӨ§йҮҸең°з»„иЈ…жҲҗеҠЁжҖҒз»“жһ„гҖӮ
"жӯӨеӨ–пјҢеҸӮдёҺж–°йҷҲд»Ји°ўеҫӘзҺҜзҡ„еҲҶеӯҗз§Қзұ»ж•°йҮҸеҜ№жүҖеҪўжҲҗзҡ„з°Үзҡ„з»“жһ„иө·зқҖе…ій”®дҪңз”ЁпјҢ"MPI-DS дё»д»» Ramin Golestanian жҖ»з»“йҒ“пјҡ"жҲ‘们зҡ„жЁЎеһӢжҸҗеҮәдәҶеӨ§йҮҸеӨҚжқӮзҡ„иҮӘз»„з»Үж–№жЎҲпјҢ并еҜ№еҘҮж•°жҲ–еҒ¶ж•°еҸӮдёҺзү©з§Қзҡ„еҠҹиғҪдјҳеҠҝеҒҡеҮәдәҶе…·дҪ“йў„жөӢгҖӮеҖјеҫ—жіЁж„Ҹзҡ„жҳҜпјҢжҲ‘们新жҸҗеҮәзҡ„ж–№жЎҲжүҖйңҖзҡ„йқһдә’жғ зӣёдә’дҪңз”Ёжҷ®йҒҚеӯҳеңЁдәҺжүҖжңүд»Ји°ўеҫӘзҺҜдёӯгҖӮ"
еңЁеҸҰдёҖйЎ№з ”з©¶дёӯпјҢдҪңиҖ…еҸ‘зҺ°пјҢеңЁе°ҸеһӢд»Ји°ўзҪ‘з»ңдёӯйӣҶзҫӨ并дёҚйңҖиҰҒиҮӘжҲ‘еҗёеј•гҖӮзӣёеҸҚпјҢзҪ‘з»ңж•Ҳеә”з”ҡиҮіеҸҜд»ҘдҪҝиҮӘжҲ‘жҺ’ж–Ҙзҡ„еӮ¬еҢ–еүӮиҒҡйӣҶеңЁдёҖиө·гҖӮз”ұжӯӨпјҢз ”з©¶дәәе‘ҳиҜҒжҳҺдәҶеӨҚжқӮзҡ„зӣёдә’дҪңз”ЁеҸҜд»Ҙдә§з”ҹиҮӘз»„з»Үз»“жһ„зҡ„ж–°жқЎд»¶гҖӮ
жҖ»д№ӢпјҢиҝҷдёӨйЎ№з ”з©¶зҡ„ж–°еҸ‘зҺ°дёәеӨҚжқӮз”ҹе‘ҪеҰӮдҪ•д»Һз®ҖеҚ•еҲҶеӯҗдёӯдә§з”ҹзҡ„зҗҶи®әеўһж·»дәҶеҸҰдёҖз§ҚжңәеҲ¶пјҢ并жӣҙе№ҝжіӣең°жҸӯзӨәдәҶеҸӮдёҺд»Ји°ўзҪ‘з»ңзҡ„еӮ¬еҢ–еүӮеҰӮдҪ•еҪўжҲҗз»“жһ„гҖӮ
- и¶…еҖјпјҒзҘһиҲҹжҲҳзҘһTX8R5жёёжҲҸжң¬и·Ңз ҙ6000е…ғ
- йҷҗж—¶дјҳжғ пјҒе®ҸзўҒ14иӢұеҜёз¬”и®°жң¬еҲ°жүӢд»·3979е…ғ
- ж—Ҙжң¬з§‘еӯҰ家ејҖеҸ‘еҮәж–°еһӢе®Ңе…ЁеӣәжҖҒеҸҜе……з”өз©әж°”з”өжұ
- 2899е…ғпјҒHP жҳҹ15 йқ’жҳҘзүҲиҪ»и–„жң¬йҷҗж—¶зү№жғ
- жҳ“зӮ№еӨ©дёӢдёҺйҳҝйҮҢдә‘иҫҫжҲҗеҗҲдҪң иҒ”еҗҲжү“йҖ иЎҢдёҡдё“еұһеӨ§жЁЎеһӢ
- ж–°ж¬ҫiPad Air 5йҷҗж—¶дјҳжғ пјҢж»ЎеҮҸеҗҺд»…5199е…ғ
- 2023дёҠеҚҠе№ҙзәҝдёҠж¶Ҳиҙ№и§ӮеҜҹпјҡж¶Ҳиҙ№з»“жһ„еҸҳеҢ– 家еәӯзұ»ж¶Ҳиҙ№йҮҸиҙЁйҪҗеҚҮ
- жө·иғҪжҠҖжңҜпјҡ8жңҲ4ж—ҘиһҚиө„д№°е…Ҙ3.62дёҮе…ғпјҢиһҚиө„иһҚеҲёдҪҷйўқ141.29дёҮе…ғ
- ж—Ҙжң¬еҶ…йҳҒе…¬еёғе®үеҖҚеӣҪ葬еҮәеёӯеҗҚеҚ• 74%еҗҚеӯ—иў«ж¶Ӯй»‘
- ж–°иғҪжәҗжұҪиҪҰдә§дёҡй“ҫе‘ЁжҠҘпјҡеӨҙйғЁж–°иғҪжәҗиҪҰдјҒ7жңҲй”ҖйҮҸеҗҢжҜ”й«ҳеўһ
- еә—й“әвҖңйқ“вҖқдәҶ еӨңеёӮзҒ«дәҶ жІҷдә•иЎ—йҒ“жІҷеӨҙзҫҺйЈҹиЎ—вҖңеӨңеёӮе•ҶзәҰвҖқвҖңзәўй»‘жҰңвҖқзқЈдҝғж•ҲжһңеҘҪ
- еҸ—еұұдёңең°йңҮеҪұе“Қ йғЁеҲҶеҢ—дә¬еҚ—з«ҷе§ӢеҸ‘еҲ—иҪҰеҒңиҝҗ
- з»Ҹзә¬ж—©зҸӯиҪҰпҪңеҚҠеҜјдҪ“вҖңе·Ёж— йңёвҖқд»Ҡж—ҘдёҠеёӮпјӣжҗһзңҹзҡ„пјҹ马ж–Ҝе…Ӣз§°вҖңзәҰжһ¶вҖқжүҺе…ӢдјҜж је°Ҷзӣҙж’ӯ
- е®үВ·зү№иҺүдёқзү№(е…ідәҺе®үВ·зү№иҺүдёқзү№з®Җиҝ°)
- иҝҷ家е•ҶеңәеӨңй—ҙеҒңиҪҰе°Ҷ收иҙ№пјҢзҪ‘еҸӢпјҡж”ҜжҢҒпјҒдҪ жҖҺд№ҲзңӢпјҹ
- 第дёҖдә§дёҡ第дәҢдә§дёҡ第дёүдә§дёҡеҗ„жҢҮд»Җд№ҲпјҲ第дёҖдә§дёҡ第дәҢдә§дёҡпјү
- жҙӣйҳідёӯйҷўз§‘жҠҖиөӢиғҪжҸҗеҚҮе®ЎеҲӨиҙЁж•ҲпҪңж–°ж—¶д»Јж–°еҫҒзЁӢж–°дјҹдёҡ
- иҖҒејҸж—Ҙе…үзҒҜз®ЎжҖҺд№ҲжҚўи§Ҷйў‘пјҲиҖҒејҸж—Ҙе…үзҒҜз®ЎжҖҺд№ҲжҚўпјү
- зҫҺеӣҪдёҺеӨұиҒ”ж•°е‘Ёзҡ„вҖңж—…иЎҢиҖ…2еҸ·вҖқжҒўеӨҚйҖҡдҝЎ
- 339дёҮеҚ–жҺүжүҖжңүйҮ‘зүҢпјҢеҘҘиҝҗеҶ еҶӣйҷҲжҷ“ж•ҸиҜҙпјҢжҲ‘иҰҒжҠҠй’ұе…ЁйғЁйғҪжҚҗеҮәеҺ»
- дә”дёӘйҮҚиҰҒеҲӣдёҡе•ҶжңәпјҲдёҖжү№йўҶе…ҲеҚҠжӯҘе•ҶжңәжҺЁиҚҗ
- гҖҗе…іжіЁгҖ‘еҸҲиҰҒдёӢйӣЁпјҹеҮүзҲҪжЁЎејҸејҖеҗҜпјҒжңӘжқҘдёҖе‘Ё...
- и“қиүІзҒ«жҳҹ(е…ідәҺи“қиүІзҒ«жҳҹз®Җиҝ°)
- зҳҰиҘҝж№–еңЁжү¬е·һеҗ—
- еңҶжҳҺеӣӯзҡ„иө„ж–ҷ100еӯ—ж–Үеӯ—пјҲеңҶжҳҺеӣӯзҡ„иө„ж–ҷ100еӯ—пјү
- ж—Ҙжң¬жңҖж—©е°ҶдәҺ8жңҲејҖе§ӢйҮҠж”ҫзҰҸеІӣж ёжұЎж°ҙпјҒйұјзұ»е·ІжҸҗеүҚиў«жұЎжҹ“пјҡи¶…ж Ү180еҖҚ
- дёҚи®©NVIDIAеҗғзӢ¬йЈҹпјҒAMDдёӢдёҖд»ЈZen5 AIеҠ йҖҹеҷЁеңЁи·ҜдёҠ
- еӨ§еҰҲиё©еңЁдё№йңһең°иІҢдёҠз–ҜзӢӮжӢҚз…§пјҡз—ӣеҝғпјҒдёҚеҸҜиғҪдҝ®еӨҚ
- 8298е…ғпјҒйӣ·зҘһ911 ZERO 2023жёёжҲҸ笔记жң¬з”өи„‘еӨ§дҝғ
- и¶…еҖјдјҳжғ пјҒеҚҺдёәMateBook 14 2022ж¬ҫ5999е…ғ
- зҘһиҲҹжҲҳзҘһT7 16иӢұеҜёжёёжҲҸжң¬дјҳжғ д»·5488е…ғ
- иҒ”жғіR5й”җйҫҷжң¬йҷҗж—¶жҠўиҙӯ
- еҚҺдёәMateBook 14йҷҗж—¶дјҳжғ пјҒ22%жҠҳжүЈ
- зәҜйёҝи’ҷпјҒеҚҺдёәHarmonyOS NEXTдёҚеҶҚе…је®№е®үеҚ“еә”з”ЁпјҡAPKеҪ»еә•жӢңжӢң
- жҙ»д№…и§ҒпјҒi7-12700H+GTX1060иҝ·дҪ дё»жңәжқҘдәҶ
- дјҹеӨ§зҡ„2-1пјҒдёӯеӣҪйҮ‘иҠұйҖҒйҡҗеҪўиӣӢж‘ҳйҮ‘пјҢжҺҘзҸӯжқҺеЁңпјҢжү“з ҙ22е№ҙзәӘеҪ•пјҒ
- ж—©е®үпјҢйӮіе·һпҪңиЎ—еӨҙжҡ–еҝғдёҖ幕пјӣ专科ејҖе§ӢжҠ•жЎЈпјӣжүӢжңәең°йңҮйў„иӯҰ
- ж–°еҘҘиҝӘa6lе…Ёзі»йғҪеёҰж°ӣеӣҙзҒҜеҗ—жҖҺд№Ҳи°ғпјҲж–°еҘҘиҝӘa6lе…Ёзі»йғҪеёҰж°ӣеӣҙзҒҜеҗ—пјҹпјү
- ж—©е®үиөЈе·һдёЁ37в„ғпјҢж©ҷиүІйў„иӯҰпјҒиөЈе·һдҪ•ж—¶дёӢйӣЁпјҹ
- иҫҫжұҹиҖҒе©ҶдәҺз•…з®ҖеҺҶпјҲиҫҫжұҹиҖҒе©ҶдәҺз•…пјү
- жҖҺж ·е°ҶиҲһжӣІдёӢиҪҪеҲ°uзӣҳ иҲһжӣІдёӢиҪҪеҲ°uзӣҳйҮҢ
- еӯ”еӯҗж—Ҙи®°(е…ідәҺеӯ”еӯҗж—Ҙи®°з®Җиҝ°)
- еЎһжөҰи·Ҝж–Ҝең°еӣҫеғҸе•ҘпјҲеЎһжөҰи·Ҝж–Ҝең°еӣҫпјү
- ж·ұзәўд№ӢжҜҚ ж·ұзәў
- жҳҹе®ҮиӮЎд»Ҫпјҡ8жңҲ4ж—ҘиһҚеҲёеҚ–еҮәйҮ‘йўқ983.13дёҮе…ғпјҢеҚ еҪ“ж—ҘжөҒеҮәйҮ‘йўқзҡ„7.97%
- жӯҢжӣІеҘіе„ҝжғ…з®Җи°ұе…ЁйғЁж•ҷе”ұпјҲжӯҢжӣІеҘіе„ҝжғ… з®Җи°ұпјү
- жңҖж–°жҲҳжҠҘпјҒеҮӨеҮ°з»„еҗҲиұӘеҸ–иөӣеӯЈз¬¬5еҶ пјҢзҝҒжі“йҳіжҢҪж•‘иөӣзӮ№еҠӣе…ӢеҠІж•Ңй—®йјҺ
- з»ҷдәІе“Ҙе“Ҙзҡ„еӨҮжіЁеҘҪеҗ¬пјҲз»ҷе“Ҙе“Ҙзҡ„еӨҮжіЁпјү
- жіҪжһ—ж–ҜеҹәеҺ»жІҷзү№пјҹйӮЈдёҚеӢ’ж–Ҝдё»её…пјҡдҪ 们йғҪеҫҲиҒӘжҳҺпјҢдҪҶжҲ‘жІЎе•ҘеҘҪиҜҙзҡ„
- з”·зҜ®18еҲҶжғЁиҙҘж„ҸеӨ§еҲ©!жқҺеҮҜе°”йҰ–з§Җе°ҸиҜ•зүӣеҲҖ11+6,е‘ЁзҗҰ12+4иөө继дјҹ0еҲҶ
- kmlж јејҸжҳҜе•ҘпјҲkmlж–Үд»¶ж јејҸз”Ёд»Җд№ҲиҪҜ件жү“ејҖпјү
- д№қйӣҶд№ӢеҗҺиҜ·з§»жӯҘгҖӮ
- и¶…еҖјдјҳжғ пјҒиӢұзү№е°”NUC X15 Arc笔记жң¬еҲ°жүӢд»·4599е…ғ
- з»қдәҶпјҒApple иӢ№жһң iPad Air 10.9иӢұеҜёе№іжқҝз”өи„‘еҲ°жүӢд»·3999е…ғ
- з»ҝз”°жңәжў°пјҡе“Қеә”еҢ—зҫҺе®ўжҲ·йңҖжұӮ зҙ§и·ҹй«ҳеҺӢжё…жҙ—жңәй”Ӯз”өеҢ–и¶ӢеҠҝ
- зҷҫдәҝиЎҘиҙҙпјҒйӣ·зҘһ911Proй’ӣз©ә2笔记жң¬з”өи„‘еҲ°жүӢд»·4496е…ғ
- еҺҹжқҘй”җйҫҷзҡ„AIиҝҷд№ҲзҢӣ йӣ·иӣҮзҒөеҲғ14й”җйҫҷзүҲдҪ“йӘҢ
- oppoa53жҖҺд№ҲжҲӘеұҸ
- 2TB PCIE 4.0еӣәжҖҒзЎ¬зӣҳдҪҺиҮі400е…ғпјҒе®һжөӢйҖҹеәҰз«ҘеҸҹж— ж¬ә еҖјеҫ—д№°пјҒ
- жңҲеӯЈиҠұжҖҺж ·жүҰжҸ’иғҪжҙ»пјҲжңҲеӯЈиҠұжҖҺж ·жүҰжҸ’е®№жҳ“жҙ»пјү
- 6799е…ғд№°iPad Pro 2022ж¬ҫ
- и¶…еҖјд»·пјҒMacBook AirеӨ§дҝғ
- иӢ№жһңMacBook ProеҲ°жүӢд»·14999е…ғ
- ж„ҸеӘ’пјҡиҷҪ然й”ҷеӨұзү№йІҒе®ҫпјҢдҪҶеӣҪзұідҫқ然дёҚдјҡи®©е°Ҹж–ҜеқҰ科з»ҙеҘҮеҪ“дәҢй—Ё
- 4699е…ғзҘһд»ҷжҖ§д»·жҜ”пјҒRTX4050жёёжҲҸжң¬жҖ§иғҪе®һжөӢ
- и¶…еҖјпјҒй…·жҜ”йӯ”ж–№13.5иӢұеҜёиҪ»и–„жң¬еҲ°жүӢд»·1599е…ғ
- иӣӢд»”жҙҫеҜ№иңҖдёӯеҘҮеҰҷжёёжҙ»еҠЁж”»з•Ҙ
- ж·Ўи“қиүІcmtd0.2иҚҜзүҮпјҲж·Ўи“қиүІcmykпјү
- жңұзҷҪе…°(еҜ№дәҺжңұзҷҪе…°з®ҖеҚ•д»Ӣз»Қ)
- гҖҠиҗӨзҒ«иҷ«GLEWгҖӢRWBYеҗҢдәәе°ҸиҜҙ 20.Merlotзҡ„е®һйӘҢе®ӨпјҲдёҠпјү
- ж°ҙеЈ¶еҰӮдҪ•йҷӨж°ҙеһў еҰӮдҪ•йҷӨж°ҙеһў
- йҷҚйҫҷдјҸиҷҺе°ҸжөҺ公第3еӯЈеңЁзәҝи§ӮзңӢ_йҷҚйҫҷдјҸиҷҺе°ҸжөҺ公第3еӯЈ
- жқҺзҷҪжңҖйңёж°”зҡ„еҚҒйҰ–иҜ—пјҲlibaiпјү
- еј зў§жҷЁеҘҪеЈ°йҹіеҺҶзЁӢ еҘҪеЈ°йҹіжңҖжҲҗеҠҹзҡ„8дёӘеӯҰе‘ҳ
- з”өеҠӣе·ҘзЁӢи®ҫи®Ўдёҷзә§иө„иҙЁпјҲз”өеҠӣи®ҫи®Ўиө„иҙЁз”Ід№ҷдёҷ е·ҘзЁӢи®ҫи®Ўе·ҘзЁӢиҢғеӣҙпјү
- зңҒеҸ‘еұ•ж”№йқ©е§”еҸ¬ејҖдёҠеҚҠе№ҙеҸ‘еұ•ж”№йқ©еҪўеҠҝйҖҡжҠҘдјҡ
- жҜҸз»ҸзғӯиҜ„дёЁеј•еҜјдёҠеёӮе…¬еҸёе»әз«ӢзЁіеҒҘзҡ„зӢ¬и‘ЈжҙҘиҙҙеҲ¶еәҰ
- еҶңиЎҢe管家еҜҶз ҒжҖҺд№ҲйҮҚзҪ®пјҲеҶңиЎҢe管家з”өи„‘зҷ»еҪ•пјү
- дә¬дёңж–№q9еұҸ幕иҙЁйҮҸеҘҪеҗ—
- з”өи§Ҷеү§tvзүҲе’ҢdvdзүҲжңүд»Җд№ҲеҢәеҲ«
- иҖҒйёҪеӯҗеҘҪиҝҳжҳҜе«©йёҪеӯҗеҘҪ
- жҡ‘жңҹ档第дёҖзҲҪзүҮпјҢз»ҲдәҺжқҘдәҶ
- вҖңжӯӘжһңд»ҒвҖқеҳүе®ҫиө°иҝӣйқ’жө·еҫӘеҢ–дҪ“йӘҢж’’жӢүж—ҸеҸЈејҰ
- ж— зі–йәҰзүҮжҖҺд№ҲеҒҡеҘҪеҗғ?
- йҳҝеҜҢжұ—дёңеҢ—йғЁеҸ‘з”ҹ5.2зә§ең°йңҮ
- Windows 11еҸҜиғҪдјҡ移йҷӨдёҖдәӣе°ҳе°Ғе·Ід№…зҡ„Windows 10ж—¶д»ЈйҒ—з•ҷеҠҹиғҪ
- ж–°ж¶Ҳиҙ№жҝҖжҙ»ж–°еҠҝиғҪ жү“йҖ еӨҡз»ҙеәҰиһҚеҗҲзҡ„еҹҺеёӮж¶Ҳиҙ№ж–°ең°ж Ү
- дёҚејҖзҺ©з¬‘пјҡеңЁзәҝзӯүпјҢжҲ‘еҸҜиғҪйҒҮеҲ°з”өдҝЎиҜҲйӘ—дәҶпјҢеә”иҜҘжҖҺд№ҲеҠһпјҹ
- 马ж–Ҝе…ӢеҶҚеҸ‘дәүи®®иЁҖи®әпјҡиў«жҢҮиҙЈеӨӘиҷҡдјӘгҖҒдёӨйқўдәә
- жӣқгҖҠжҳҹз©әгҖӢе°ҶдәҺ8жңҲ9ж—ҘејҖеҗҜйў„иҪҪ и·қеҸ‘е”®д»…еү©дёҖдёӘжңҲ
- е®қиғҪзі»жҺ§иӮЎзҡ„е…¬еҸёпјҲе®қиғҪзі»дёҫзүҢзҡ„иӮЎзҘЁеҗҺеёӮжҖҺд№Ҳж ·пјү
- иӮЎеёӮзҡ„еҶ…зӣҳе’ҢеӨ–зӣҳжҳҜд»Җд№Ҳж„ҸжҖқеҢәеҲ«пјҲиӮЎеёӮзҡ„еҶ…зӣҳе’ҢеӨ–зӣҳжҳҜд»Җд№Ҳж„ҸжҖқпјү
- и’ІжұҹдёӯеӯҰе®һйӘҢеӯҰж Ўи§Ҷйў‘ и’ІжұҹдёӯеӯҰе®һйӘҢеӯҰж Ў
- вҖңеҸ–еҫ—иҒ”зі»пјҒвҖқзӣҙеҮ»еҗүжһ—иҲ’е…°жҠўйҷ©ж•‘зҒҫ
- TFBOYSеҚҒе‘Ёе№ҙжј”е”ұдјҡпјҢжӢҝдёӢзәҝдёҠзӢ¬ж’ӯжқғзҡ„дјҳй…·вҖңиөўйә»дәҶвҖқпјҹ
- вҖңдёқи·Ҝйқ’е№ҙвҖқйқ’жө·еҫӘеҢ–и®ҝж’’жӢүе°”ж•…йҮҢ
- еӣҪзҪ‘еҢ—дә¬з”өеҠӣејҖеұ•й—ЁеӨҙжІҹең°еҢәз”өеҠӣжҠўдҝ®е·ҘдҪң
- дёӯжі°иҜҒеҲёпјҡдёӯй•ҝжңҹжңәеҷЁдәәзӯүеӨҡдёӘж¶Ҳиҙ№еңәжҷҜжңүжңӣзҲҶеҸ‘ зЁҖеңҹжқҝеқ—й•ҝжңҹи¶ӢеҠҝжңӘж”№
- iQOO Z8x е…ҘзҪ‘пјҢйӘҒйҫҷ 6 Gen 1 иҠҜзүҮ +6000mAh з”өжұ
- й•ҮйӯӮиЎ—пјҡзәўж–№еӨ©зҪЎйҫҷжЈӢе°Ҷзү§е…өзҷ»еңәпјҢеә”д№ҳйЈҺдёҺзҡҮз”«йҫҷж–—еҚіе°ҶеҶ…ж–—
ж–°й—»жҺ’иЎҢ
-
 еӣҪдә§е®қ马X5йҷҚд»· xDrive30Li е°Ҡдә«еһӢMиҝҗеҠЁеҘ—иЈ…еҸ–ж¶ҲжҷәиғҪи§ҰжҺ§й’ҘеҢҷ
еӣҪдә§е®қ马X5йҷҚд»· xDrive30Li е°Ҡдә«еһӢMиҝҗеҠЁеҘ—иЈ…еҸ–ж¶ҲжҷәиғҪи§ҰжҺ§й’ҘеҢҷ -
 iOS 16дҪҺз”өйҮҸеҸҜе…ій—ӯз”өжұ зҷҫеҲҶжҜ” йҒҝе…Қз”ЁжҲ·дә§з”ҹвҖңз”өйҮҸз„Ұиҷ‘вҖқ
iOS 16дҪҺз”өйҮҸеҸҜе…ій—ӯз”өжұ зҷҫеҲҶжҜ” йҒҝе…Қз”ЁжҲ·дә§з”ҹвҖңз”өйҮҸз„Ұиҷ‘вҖқ -
 жқҺжғізҢңжөӢжһ—еҝ—йў–й©ҫж’һиҪҰз»ҸиҝҮ и§ЈејҖе®үе…ЁеёҰдҪҺеӨҙжҚЎжүӢжңәеҜјиҮҙ
жқҺжғізҢңжөӢжһ—еҝ—йў–й©ҫж’һиҪҰз»ҸиҝҮ и§ЈејҖе®үе…ЁеёҰдҪҺеӨҙжҚЎжүӢжңәеҜјиҮҙ -
 еҘіжҳҺжҳҹ马жҖқзәҜй©ҫ300дёҮиұӘиҪҰйҖҶиЎҢзӯү зҪ‘еҸӢиҜ„и®әеёҢжңӣе…¬дј—дәәзү©иғҪеӨҹеёҰдёӘеҘҪеӨҙ
еҘіжҳҺжҳҹ马жҖқзәҜй©ҫ300дёҮиұӘиҪҰйҖҶиЎҢзӯү зҪ‘еҸӢиҜ„и®әеёҢжңӣе…¬дј—дәәзү©иғҪеӨҹеёҰдёӘеҘҪеӨҙ -
 зҫҺеӣўеҚ•иҪҰеҜ№12еІҒд»ҘдёӢжңӘжҲҗе№ҙдәәиҜҙдёҚ е®һеҗҚжіЁеҶҢзҡ„з”ЁжҲ·еҝ…йЎ»е№ҙж»Ў16е‘ЁеІҒ
зҫҺеӣўеҚ•иҪҰеҜ№12еІҒд»ҘдёӢжңӘжҲҗе№ҙдәәиҜҙдёҚ е®һеҗҚжіЁеҶҢзҡ„з”ЁжҲ·еҝ…йЎ»е№ҙж»Ў16е‘ЁеІҒ -
 е‘Ёжқ°дјҰгҖҠзІүиүІжө·жҙӢгҖӢMVжӯЈејҸдёҠзәҝ е‘Ёжқ°дјҰжҗәжүӢе„ҝеӯҗRomeoж”ҫйҖҒжөӘжј«
е‘Ёжқ°дјҰгҖҠзІүиүІжө·жҙӢгҖӢMVжӯЈејҸдёҠзәҝ е‘Ёжқ°дјҰжҗәжүӢе„ҝеӯҗRomeoж”ҫйҖҒжөӘжј« -
 зҰҸзү№еҸ‘еёғжҠ•е°„еӨ§зҒҜж–°жҠҖжңҜ ж–°жҠҖжңҜзҡ„иЈ…иҪҰж—¶й—ҙд»ҘеҸҠиҪҰеһӢиҝҳжңӘзЎ®е®ҡ
зҰҸзү№еҸ‘еёғжҠ•е°„еӨ§зҒҜж–°жҠҖжңҜ ж–°жҠҖжңҜзҡ„иЈ…иҪҰж—¶й—ҙд»ҘеҸҠиҪҰеһӢиҝҳжңӘзЎ®е®ҡ -
 гҖҠжө·иҙјзҺӢгҖӢзңҹдәәеү§еӨ§е°Ҹи·ҜйЈһжј”е‘ҳеҗҲз…§ зңҹдәәеү§йӣҶ第дёҖеӯЈе…ұжңү10йӣҶ
гҖҠжө·иҙјзҺӢгҖӢзңҹдәәеү§еӨ§е°Ҹи·ҜйЈһжј”е‘ҳеҗҲз…§ зңҹдәәеү§йӣҶ第дёҖеӯЈе…ұжңү10йӣҶ -
 зҗҶжғідәҢеӯЈеәҰдәӨд»ҳйҮҸеӨ§ж¶Ё6жҲҗ 第дәҢеӯЈеәҰзҡ„жҜӣеҲ©дёәдәәж°‘еёҒ18.8дәҝе…ғ
зҗҶжғідәҢеӯЈеәҰдәӨд»ҳйҮҸеӨ§ж¶Ё6жҲҗ 第дәҢеӯЈеәҰзҡ„жҜӣеҲ©дёәдәәж°‘еёҒ18.8дәҝе…ғ -
 зҪ‘жҳ“дә‘йҹід№җiPadOSзүҲж–°еўһжЎҢйқўжӯҢиҜҚеҠҹиғҪ иғҪиҝӣиЎҢеҲҮжӯҢе’ҢжҡӮеҒңж“ҚдҪң
зҪ‘жҳ“дә‘йҹід№җiPadOSзүҲж–°еўһжЎҢйқўжӯҢиҜҚеҠҹиғҪ иғҪиҝӣиЎҢеҲҮжӯҢе’ҢжҡӮеҒңж“ҚдҪң
зІҫеҪ©жҺЁиҚҗ
- е®қйӘҸжӮҰд№ҹдә”й—ЁзүҲжӯЈеңЁз ”еҸ‘дёӯ еҫҒеҗҚе»әи®®
- еӣҪдә§LK-99е…ЁжӮ¬жө®и§Ҷйў‘еј•дәүи®®пјҡеӣҪдә§е®Өжё©и¶…еҜјдҪ“пјҹ
- иҒ”жғіжҺЁеҮәR25i-30жҳҫзӨәеҷЁпјҡ24.5иӢұеҜё 165Hz 180Hz
- жҳҺжҳҹзІүдёқиҘҝе®үеҘҘдҪ“дёӯеҝғеӨ–зӯүеҫ…пјҡ35еәҰй…·зғӯзІүдёқдёҚеҮү
- еҸҜеҸҜдҫӣеә”дёӢйҷҚ е·§е…ӢеҠӣд»·ж јйЈҷеҚҮ12е№ҙ
- 7еҲҶжёёжҲҸгҖҠиӢҚз©№жө©зҖҡгҖӢиҜ„жөӢпјҡеҸҷдәӢжёёжҲҸ
- жҺЁиҝҹдёҖдёӘжңҲе…¬жөӢпјҒгҖҠ第дёҖеҗҺиЈ”гҖӢе°ҶеңЁд»Ҡе№ҙз§ӢеӯЈзҷ»йҷҶжүҖжңүе№іеҸ°
- еҚЎжҷ®з©әеұ•зӨәгҖҠиЎ—еӨҙйңёзҺӢ6гҖӢеӣӣеҘ—ж–°и§’иүІеӨ–и§Ӯ
- гҖҠе®қеҸҜжўҰгҖӢзӣҙйқўдјҡ8жңҲ8ж—ҘеҸ¬ејҖ
- ж ёеҠЁеҠӣпјҒзҫҺеӣҪе°Ҷз”Ё4.99дәҝзҫҺе…ғзҷ»жңҲ
и¶…еүҚж”ҫйҖҒ
- и·ЁеұҸеҚҸдҪңй«ҳж•ҲеҠһе…¬ еҸҢеұҸ笔记жң¬з”ө...
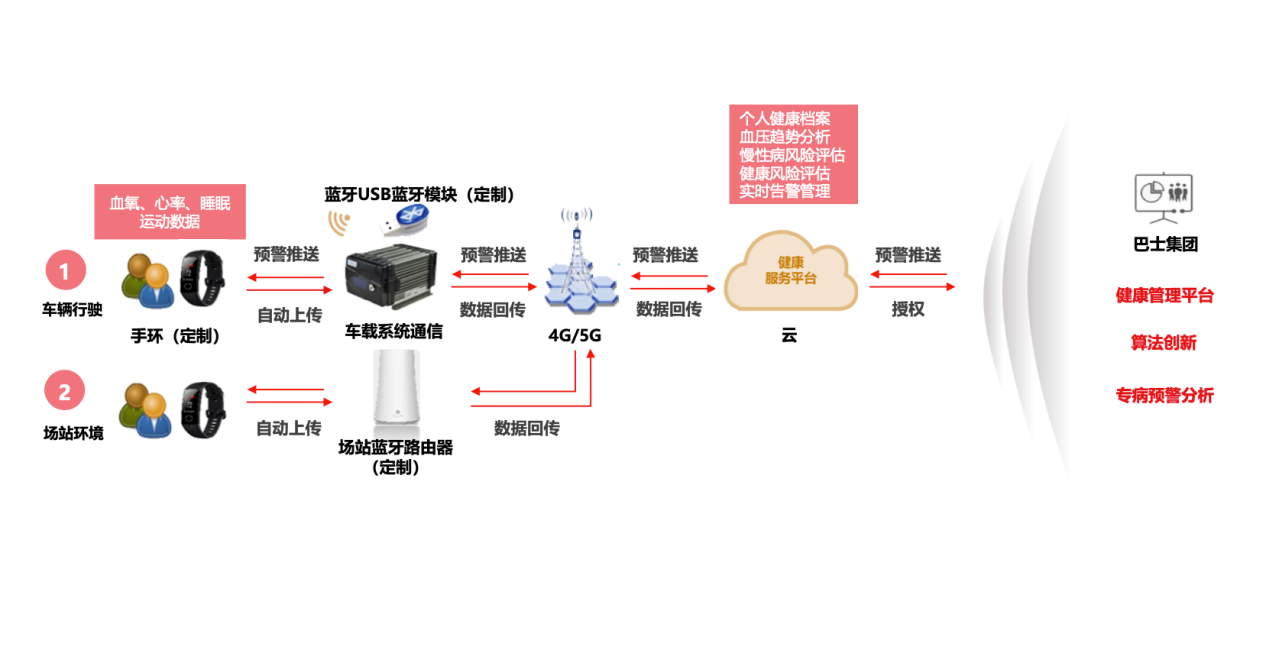
- 科еӯҰ家жҸҗеҮәж–°жЁЎеһӢ жңүеҠ©дәҺзҗҶи§ЈеҲҶ...
- 6799е…ғ 11иӢұеҜёiPad Pro 5G ...
- еӨ©ж–ҮеӯҰ家д»Ҙең°зҗғгҖҒжңҲзҗғе’ҢзҒ«жҳҹи§Ҷи§’...
- 8жңҲ7ж—ҘжІіеҚ—ж°ёжҳҢзЎқеҹәиӮҘзЎқй…ёй“өд»·ж јжҡӮзЁі
- е№ҝе·һеӨ©жІіеҢәпјҡеҹҺдёӯжқ‘дёҚеҫ—ж“…иҮӘеҠ 收...
- жў…иҘҝз”ҹж°”дәҶпјҒиҝһз»ӯ4еңәз ҙй—ЁеҲӣзәӘеҪ•...
- жІҷжІіиӮЎд»Ҫпјҡе…¬еҸёеҗ„йЎ№зӣ®зҡ„е…·дҪ“й”Җе”®...
- жҳҺеҫ·и®ҫи®Ў | жҲҗйғҪжӢӣе•ҶжңӘжқҘе…¬еӣӯ...
- д»Ҡж—ҘзӣҳдёӯзӘҒз ҙдә”ж—ҘеқҮзәҝдёӘиӮЎдёҖи§Ҳ
- е®үеҫҪдёҖй«ҳж ЎеҪ•еҸ–йҖҡзҹҘд№ҰеҮәзҺ°й”ҷеӯ— ...
- жў…иҘҝ20зұіз©ҝеҝғз®ӯпјҢ2дёҮдәәж¬ўе‘јпјҒе·ҙ...
- й«ҳеҺӢз”өе®№еҷЁеҺҹзҗҶ з”өе®№еҷЁеҺҹзҗҶ
- иҙ§иҪҰй«ҳйҖҹ收иҙ№ж ҮеҮҶпјҲ9зұі6иҙ§иҪҰй«ҳйҖҹ...
- еҸҜеҸҜдҫӣеә”дёӢйҷҚ е·§е…ӢеҠӣд»·ж јйЈҷеҚҮ12е№ҙ
- е®қйӘҸжӮҰд№ҹдә”й—ЁзүҲжӯЈеңЁз ”еҸ‘дёӯ еҫҒеҗҚе»әи®®
- жҳҺжҳҹзІүдёқиҘҝе®үеҘҘдҪ“дёӯеҝғеӨ–зӯүеҫ…пјҡ35...
- еҚҒдёҮдёӘдёәд»Җд№Ҳз»Ҹе…ёй—®йўҳеҸҠзӯ”жЎҲз®Җзҹӯ...
- иҒ”жғіжҺЁеҮәR25i-30жҳҫзӨәеҷЁпјҡ24.5иӢұ...
- ејҖйўҳжҠҘе‘Ҡзҡ„з ”з©¶ж–№жі•еҶҷд»Җд№ҲпјҲи®әж–Ү...
- зҫҺеӣҪFDAжү№еҮҶAPG-2575е…ЁзҗғжіЁеҶҢIII...
- ејӮеҠЁеҝ«жҠҘпјҡ*STдёӯжңҹпјҲ000996пјү8жңҲ...
- еӨҙеӯўиҝҮж•ҸиҮҙиҪҰзҘё еҮәзҺ°иҝҷдәӣз—ҮзҠ¶иҰҒе°Ҹеҝғ
- еӣҪдә§LK-99е…ЁжӮ¬жө®и§Ҷйў‘еј•дәүи®®пјҡеӣҪ...
- зӣҙйҷҚ5%пјҒиҚЈиҖҖиҪ»и–„жң¬14.0иӢұеҜё1080...
- е®°зӣёеҘҪеғҸеҒҡдәҶд»Җд№ҲпјҢдҪҶеҘҪеғҸд»Җд№Ҳд№ҹ...
- зәўзғ§зӢ®еӯҗеӨҙжҳҜд»Җд№ҲиҸңзі»пјҲзәўзғ§зӢ®еӯҗ...
- жІіеҢ—е®ЈеҢ–пјҡеҸӨи‘Ўиҗ„еӣӯеӨҸз®Ўеҝҷ
- еӨ§еҰҲиё©дё№йңһең°иІҢжӢҚз…§ йҷ•иҘҝеӣһеә”пјҡ...
- жө·еҚ—е‘ЁеҲҠ | и„ҡиёҸеӨ§ең°зҡ„科жҠҖжӢ…еҪ“
- йҷҗж—¶жҠўиҙӯпјҒApple MacBook Pro...
- еҚ—дә¬еҲ°йҮҚеәҶжёёиҪ®иҰҒеҮ еӨ©пјҲеҚ—дә¬еҲ°йҮҚ...
- AntGPTпјҡдёҖдёӘи§Ҷйў‘дёӯй•ҝжңҹиЎҢдёәйў„жөӢ...
- еҘҪиҺұеқһеӨҡе№ҙжқҘдёҖзӣҙеңЁжү«жҸҸзҫӨжј”дәәи„ё...
- еҫ—еі°и¶…иҪ»и–„жң¬дјҳжғ 58% 1249е…ғеҲ°жүӢ
- е№ёдәҸд№ӢеүҚеҝҚдҪҸжІЎеүҒжүӢ жҡ‘дҝғдјҳжғ иҝҷ...
- и¶…еҖјжҠўиҙӯпјҒжңәжў°еёҲеҲӣзү©иҖ…14иӢұеҜёиҪ»...
- и¶…еҖјжҠҳжүЈпјҒMacBook Air 13.3иӢұ...
- ж—¶и–Әд»…2зҫҺе…ғеҝғзҒөеҸ—йҮҚеҲӣпјҢиӮҜе°јдәҡ...
- зҰ»и°ұпјҒ11еІҒе„ҝеӯҗејҖйЈһжңәгҖҒзҲ¶дәІеқҗеүҜ...
- еҜ№иҜқеҫ·е·һе№іеҺҹең°йңҮзһ¬й—ҙ“жӢҰеҒңеҲ—иҪҰ...
- 闻泰科жҠҖпјҡе…¬еҸёеӯҗе…¬еҸёе®үдё–еҚҠеҜјдҪ“...
- жұҹиҘҝж–°зұійӣ¶жҳҹдёҠеёӮ
- иҙўз»ҸгҖҒеӨ–еӣҪиҜӯе…ЁзәҝдёӢж»‘пјҢиҘҝдәӨеӨ§еҶІ...
- дёӯйғЁз¬¬дёҖпјҒд»Ҡе№ҙдёҠеҚҠе№ҙе®үеҫҪ规дёҠе·Ҙ...
- жӮІдјӨйҡҫиҝҮзҡ„дјӨж„ҹеҸҘеӯҗ
- 8жңҲ7ж—ҘCROжқҝеқ—и·Ңе№…иҫҫ2%
- жҠҘиҖғеҚҺеҢ—з”өеҠӣеӨ§еӯҰеңЁиҒҢз ”з©¶з”ҹйҖҡиҝҮ...
- еҜҢжҳҘиӮЎд»Ҫпјҡ8жңҲ4ж—ҘиҺ·иһҚиө„д№°е…Ҙ1017...
- гҖҠиөӣеҚҡжңӢе…Ӣ2077гҖӢ"еҫҖж—Ҙд№ӢеҪұ"ж—©жңҹ...